r
basics
calculator
significance-tables
sig-tables
R als Nachschlagewerk für statistische Tabellen
Allgemeines
Jede Verteilung hat einen Namen z. B. t oder f oder chisq.
Durch Voranstellen von
- d Dichte (density) Ordinate, y
- p Wahrscheinlichkeit (probability, CDF, Cumulative Distribution Function), Fläche unter der Kurve
- q Quantil (Punkt-Werte z. B. für bestimmte Alpha-Grenzen) Abszisse, x
- r Erzeugt entsprechend verteilte Daten z. B. für Simulationen (random deviates)
kann man verschiedene, auf derselben Tabelle beruhenden Werte ausgeben lassen.
Beispiele:
# F-krit für Zähler-df=100, Nenner-df=10 Freiheitsgrade bei Alpha = 0.05 qf(0.05, 100, 10, lower.tail = FALSE) # t-krit für df=22 Freiheitsgrade bei Alpha = 0.05 einseitig qt(0.05, 22, lower.tail = FALSE) # t-krit für df=55 Freiheitsgrade bei Alpha = 0.01 zweiseitig # Achtung, für zweiseitige Fragestellung wird der Alpha-Wert halbiert qt(0.005, 55, lower.tail = FALSE) # Chi-Quadrat für df=100 Freiheitsgrade bei Alpha = 0.01 qchisq(0.01, 100) # Binomialverteilung # Wahrscheinlichkeit, bei 3 mal Münzwurf zwei mal Zahl zu bekommen # n=3, k=2, p=0.5 dbinom(2, 3, 0.5) # Standardnormalverteilung # z-Wert der die unteren 2.5% der Fläche unter der Verteilung abschließt qnorm(.025, 0, 1, lower.tail=T) # z-Transformation von IQ-Werten # iq = 95, MW=100, sd=15 # z = (x - MW)/sd # welchen Prozentrang hat dieser Proband? z <- (95 - 100)/15 pnorm(z, 0, 1) # Proband hat Prozentrang von 37, also 37% der Pop sind weniger intelligent als er # Erzeugen einer normalverteilten Gruppe von 1000 IQs iqs <- rnorm(1000, 100, 15) # und zur Kontrolle ansehen hist(iqs) qqnorm(iqs) qqline(iqs)
Es gibt die folgenden Tabellen mit den jeweiligen Parametern
| Distribution | R name | additional arguments |
|---|---|---|
| beta | beta | shape1, shape2, ncp |
| binomial | binom | size, prob |
| Cauchy | cauchy | location, scale |
| chi-squared | chisq | df, ncp |
| exponential | exp | rate |
| F | f | df1, df2, ncp |
| gamma | gamma | shape, scale |
| geometric | geom | prob |
| hypergeometric | hyper | m, n, k |
| log-normal | lnorm | meanlog, sdlog |
| logistic | logis | location, scale |
| negative | binomial | nbinom size, prob |
| normal | norm | mean, sd |
| Poisson | pois | lambda |
| Student's t | t | df, ncp |
| uniform | unif | min, max |
| Weibull | weibull | shape, scale |
| Wilcoxon | wilcox | m, n |
data
dataframe
dataframe
Data-Frames
# DataFrames
############
# ist Liste, deren Komponenten Vectoren,
# factoren, numerische Matrizen Listen oder andere dataframes sind
# Enspricht am ehesten Datenstrukturen in anderen Statistikpaketen
# Spalten können z. B. Vectoren mit Namen entsprechen
# und können unter diesen Namen angesprochen werden
# data.frame generieren 'zu Fuß'
data <-data.frame(
nr=c(1,2,3,4,5),
geschl=c('w','m','m','w','w'),
spass=c(10, 8, 7, 9, 3))
# Denselben data.frame generieren aus Einzelvectoren
nr=c(1,2,3,4,5),
geschl <- c('w','m','m','w','w')
spass <- c(10, 8, 7, 9, 3)
data <- data.frame(nr, geschl, spass)
# Datenspalte anhängen
data['neue.spalte'] <- c(101,102,103,104,105) # Spaltenname darf noch nicht existieren
# Spaltennamen ausgeben
names(data) # prima bei sehr großen Datenobjekten um z. B. Position einer Spalte festzustellen
# Dataframes können aus externen Dateien eingelesen werden
# die z. B. mit Spreadsheets (Excel, etc.) erstellt wurden
# erste Zeile kann die Spaltennamen enthalten
# die werden dann in Vectoren diesen Namens konvertiert
# Datenfiles
# read.table und read.delim erzeugen data.frames
# sind Textfiles, default: Tab-delimited
# bequemstes Einlesen: Tab als Delimiter, leere Zelle als missing (NA)
my.data <- read.delim("http://www.psych.uni-goettingen.de/r/files/and_then.dat")
# Editieren von Dataframes
fix(dataframe.name)
# öffnet dataframe und stellt es spreadsheetartig dar und ermöglicht editieren
# wenn nicht direkt beim fix-Befehl ein neuer (oder derselbe) Name zugewiesen wird,
# gehen die Änderungen verloren
my.data <- read.delim("http://www.psych.uni-goettingen.de/r/files/and_then.dat")
my.data <- fix(my.data) # my.data wird überschrieben
my.data.new <- fix(my.data) # my.data bleibt unverändert
# Dataframes können aktiviert werden
# d. h. man kann auf die Komponenten (Datenvectoren) über deren Namen zugreifen
# ohne die Schreibweise dataframename$spaltenname
data <-data.frame(
nr=c(1,2,3,4,5),
geschl=c('w','m','m','w','w'),
spass=c(10, 8, 7, 9, 3))
spass
# ergibt Error
attach(data)
# legt 'data' in den 'Suchpfad'
geschl
# zeigt Vector 'geschl' des dataframe 'data'
spass
# zeigt Vector 'spass' des dataframe 'data'
detach(data)
# nimmt 'data' aus dem 'Suchpfad'
spass
# ergibt Error
data$spass
# zeigt Vector 'spass' des dataframe 'data'
# Löschen aus data.frame
data <-data.frame(
nr=c(1,2,3,4,5),
muell = c(1,1,1,1,1),
geschl=c('w','m','m','w','w'),
spass=c(10, 8, 7, 9, 3))
data # mit Muell Spalte
data <- data.frame(nr = data[,1], data[,3:4])
data # jetzt ohne Muell-Spalte
# vp 3 löschen
data <- data[data$nr != 3,]
data # data ohne vp 3
# invertieren: Columns werden zu rows und umgekehrt
data # original
t(data) # invertiert
# einen Faktor in Abhängigkeit von einer anderen Spalte (Variable) generieren
# einlesen
my.data <- read.delim("http://www.psych.uni-goettingen.de/r/files/data_befinden_gewicht.txt")
# spalte gruppe anhängen, default 0
my.data['gruppe'] <- 0
my.data$gruppe[my.data$gew > 100] = 2 # gewicht größer 100: Gruppe = 2
my.data$gruppe[my.data$gew <= 100] = 1 # gewicht kleiner gleich 100: Gruppe = 1
my.data$gruppe <- factor(my.data$gruppe) # und nun zum Faktor deklarieren und die Spalte überschreiben
#? sortieren, umsortieren
# Data Frame in Datei wegschreiben (tab-delimited)
write.table(my.data, "", sep="\t")
# wenn die String-Variablen und die Variablennamen in Quotes ("...") stören:
write.table(my.data, "", sep="\t", quote=F)
# wenn die Fallnummern stören bzw. das Verrutschen der Variablennamen(Spaltennamen):
write.table(my.data, "", sep="\t", quote=F, row.names=F)
# sortieren, umsortieren von data.frame
data <-data.frame(
nr=c(1,2,3,4,5,6),
muell = c(1,2,5,9,10,1),
geschl=c('w','m','m','w','w','m'),
spass=c(10, 8, 7, 9, 3, 9))
# sortieren nach Spalte data$spass
data.sortet <- data[order(data$spass),]
# und zeigen
data
data.sortet
# sortieren nach zwei Spalten: data$spass und sekundär nach data$muell
data.sortet <- data[order(data$spass, data$muell),]
und zeigen
data.sortet
slicing
slicing
Slicing: Zugriff auf Teile von Datenobjekten
bei Vektoren:
# zwei Datenvektoren: sex <- c(1,2,2,1,1,1,2,2,2,1,2,2,1,1,1,2,1) age <- c(15, 17, 22, 32, 12, 18, 24, 34, 23, 22, 29, 15, 17, 20, 21, 23, 19) # die ersten 5 Alterswerte: age[1:5] # die Werte 2, 5, 7-10 des Vektors age[c(2, 5, 7:10)] # Bedingter Zugriff (Filter) # die Werte unter 20 age[age < 20] # Zugriff kann auch erfolgen über anderen Vektor und Bedingung hieraus (Filter aufgrund anderer Daten) # alle Werte von age für sex = 1 age[sex == 1] # auch kombinierte Bedingungen sind möglich # alle Werte von age > 10 und sex = 2 age[sex == 2 & age > 10]
Slices bei Dataframes
Zugriff auf Spalten
# Beispiel data.frame generieren
data <-data.frame(
nr=c(1,2,3,4,5),
geschl=c('w','m','m','w','w'),
spass=c(10, 8, 7, 9, 3))
# Zugriff auf einzelne Spalten
# Über den Spaltennamen
# Name data.frame, ein $-Zeichen, Name der Spalte
data$geschl
# oder
data['geschl']
# oder
data[,1]
Teilmatrizen, Teil-Dataframes
in der Slice-Klammer mit Komma getrennt Zeilenangabe, Spaltenangabe '[Zeilen, Spalten]'
fehlt eine der beiden Angaben, gilt es für alle
data <-data.frame(
nr=c(1,2,3,4,5),
geschl=c('w','m','m','w','w'),
spass=c(10, 8, 7, 9, 3))
# erste Zeile (Vp 1) zweite Variable (Geschlecht) ausgeben:
data[1,2]
# erste Zeile/Vp ausgeben, alle Variablen
data[1,]
# das geht auch mit dem bedingten Zugriff
# alle Frauen
data[data$geschl == 'w',]
# alle Frauen mit Spass < 10
data[data$geschl == 'w' & data$spass < 10,]
datafiles
datafiles
Input-/Outputmöglichkeiten in R
Data Frames
Data-Frames haben wegen ihrer Bedeutung zum Datenaustausch mit anderen Programmen eine hohe Wichtigkeit und eigene Befehle.
Einlesen eines Datenobjektes 'my.data' aus einer externen Datei im tab-delimited Format.
# Einlesen einer Datei 'and_then.dat' von einem Laufwerk p:
my.data <- read.delim("p:/and_then.dat")
# Einlesen einer Datei irgendwo im Web über eine URL
my.data <- read.delim("http://www.psych.uni-goettingen.de/r/files/and_then.dat")
Export bzw. wegschreiben eines Datenobjektes 'my.data':
# Data Frame in Datei wegschreiben (default settings)
write.table(my.data, "p:/my_data.dat")
# Data Frame in Datei wegschreiben (tab-delimited)
write.table(my.data, "p:/my_data.dat", sep="\t")
# wenn die String-Variablen und die Variablennamen in Quotes ("...") stören:
write.table(my.data, "p:/my_data.dat", sep="\t", quote=F)
# wenn die Fallnummern stören bzw. das Verrutschen der Variablennamen(Spaltennamen):
write.table(my.data, "p:/my_data.dat", sep="\t", quote=F, row.names=F)
Spreadsheet-Dateien (Excel, Calc, ...)
Spreadsheet-Dateien am besten exportieren und dann mit read.table() oder read.csv() einlesen.
Der Export aus R für Spreadsheet-Dateien geht mit write.table() bzw. write.csv().
graphics
index
R als Grafikprogramm
Man kann R auch nur als Grafikprogramm benutzen.
In R werden Grafiken programmiert.
Eine Sammlung von Beispielen.
bar-graphs
index
R Säulendiagramme
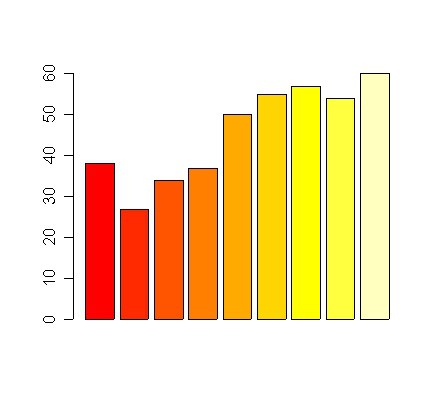
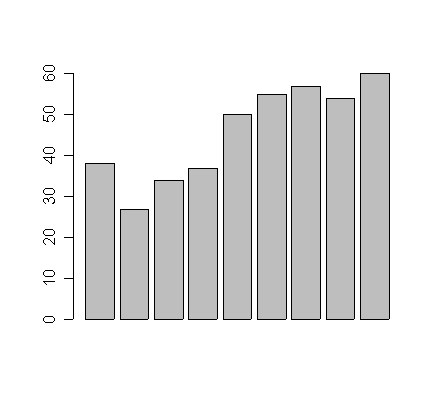
# Befinden, T-Werte (Interventions-, Kontrollgruppe, t=Messzeitpunkte) b.t <- c(38, 27, 34, 37, 50, 55, 57, 54, 60) b.w <- c(40, 38, 41, 40, 37, 32, 33, 35, 33) t <- c( 1, 2, 3, 7, 11, 15, 19, 50, 90) # Einfach nur bunte Balken barplot(b.t)
# Farbliche Festlegung eine Farbe barplot(b.t, col='grey') # colors() zeigt alle verfügbaren Farbnamen an (über 650)
# Fülleffekte # werden erreicht durch shading lines die eine Dichte und eine Richtung haben # werden solange aneinandergehängt bis die Anzahl der Säulen erreicht ist barplot(b.t, col='white', density=c(2,8),angle=c(45,180))
# Farben der Säulen explizit einzeln festlegen
barplot(c(50,70,100,80,60),col=c('white','green','red','green','white'))

# Farbliche Festlegung, die ersten drei schwarz, dann grau
barplot(b.t, col=c(rep('black',3), rep('grey',6)))
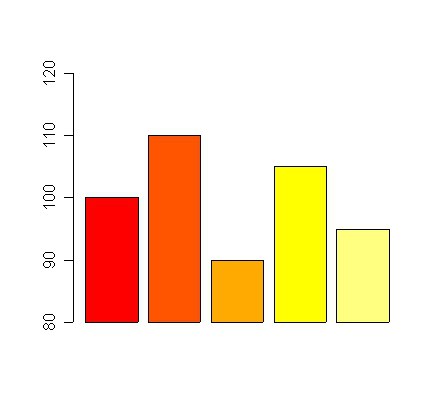
# Bars beschriften dd <- c(100,110,90,105,95) barplot(dd,names.arg=c('normal','viel','nichts','gut','wenig'))
# Y-Achse in auf bestimmte Ausmaße festlegen # xpd=F verhindert, dass über die Achse hinausgezeichnet wird dd <- c(100,110,90,105,95) barplot(dd, ylim = c(80,120),xpd=FALSE)
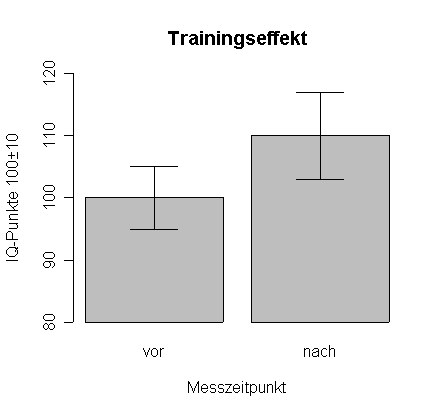
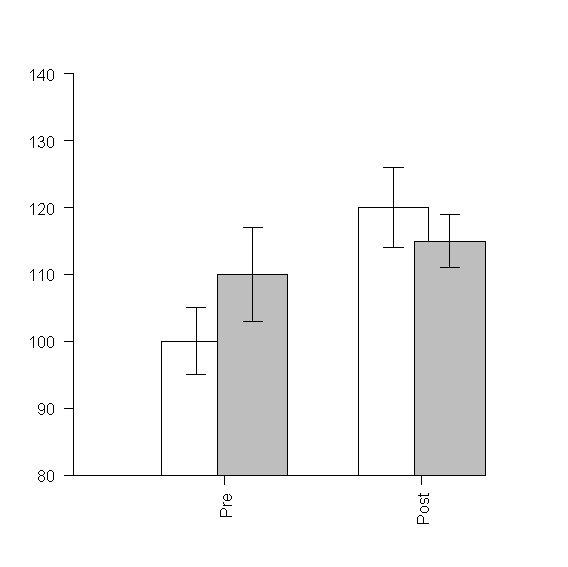
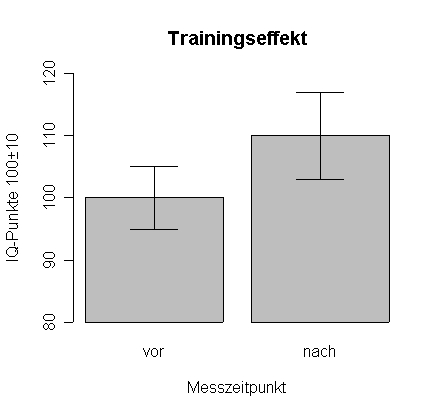
# Streuungsinformationen hinzufügen zu Barplots z. B. ein T-Test
# Prinzip: Pfeile hinzufügen mit 90-Grad Spitze
mw <- c(100,110)
se <- c(5,7)
xx <- barplot(mw,
ylim=c(80,120), # y-Achsenausdehnung festlegen
xpd=F, # Grafik darf nicht über Achsen weggehen
col='grey', # Farbe festlegen
names.arg=c('vor','nach'), # Säulen beschriften
main='Trainingseffekt', # Titel hinzufügen
xlab='Messzeitpunkt', # x-Achsen Label
ylab='IQ-Punkte 100±10' # y-Achsen Label
)
# in xx werden die x-Koordinaten gespeichert
# xpd=F verhindert, daß Säulen unter die x-Achse 'sinken'
arrows(xx[1],mw[1],xx[1],mw[1] + se[1], angle=90)
arrows(xx[1],mw[1],xx[1],mw[1] - se[1], angle=90)
arrows(xx[2],mw[2],xx[2],mw[2] + se[2], angle=90)
arrows(xx[2],mw[2],xx[2],mw[2] - se[2], angle=90)
# Um den Umgang zu erleichtern als Funktion
t.bild <- function(mw, se, main='', col=c('white','white'), ylab='', xlab='', names=c('','')){
xx <- barplot(mw,
max.y <- round((max(mw) + max(se)) * 1.10),
ylim=c(0,max.y), # y-Achsenausdehnung festlegen
xpd=F, # Grafik darf nicht über Achsen weggehen
col=col, # Farbe festlegen
names.arg=names, # Säulen beschriften
main=main, # Titel hinzufügen
xlab=xlab, # x-Achsen Label
ylab=ylab # y-Achsen Label
)
arrows(xx[1],mw[1],xx[1],mw[1] + se[1], angle=90)
arrows(xx[1],mw[1],xx[1],mw[1] - se[1], angle=90)
arrows(xx[2],mw[2],xx[2],mw[2] + se[2], angle=90)
arrows(xx[2],mw[2],xx[2],mw[2] - se[2], angle=90)
}
# und aufrufen
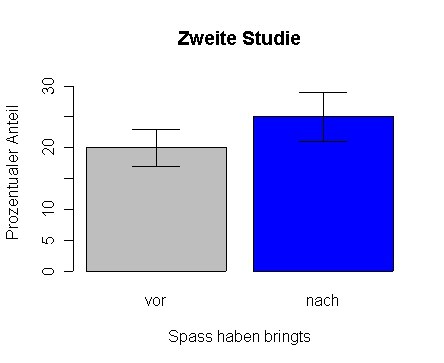
t.bild(c(20,25), c(3,4))
t.bild(c(20,25), c(3,4), col=c('grey', 'blue'), main='Zweite Studie', xlab='Spass haben bringts', ylab='Prozentualer Anteil', names=c('vor','nach'))
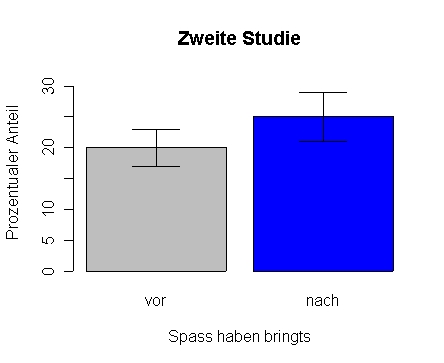
bar-graphs-grouped
index
R Säulendiagramme
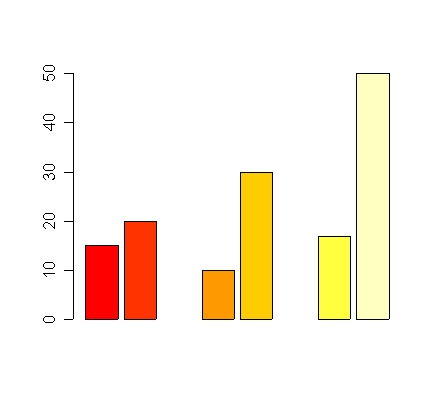
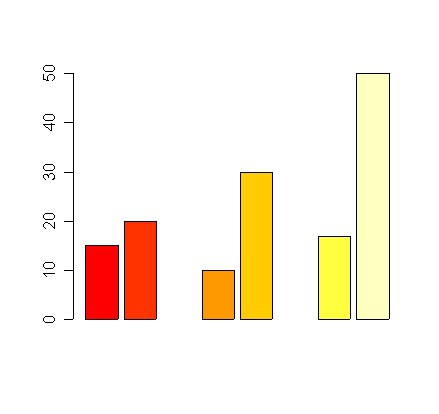
# gruppierte Säulendiagramme - barplots # Gruppierte Balken durch eingestreute fehlende Werte barplot(c(15,20,NA,10,30,NA,17,50))
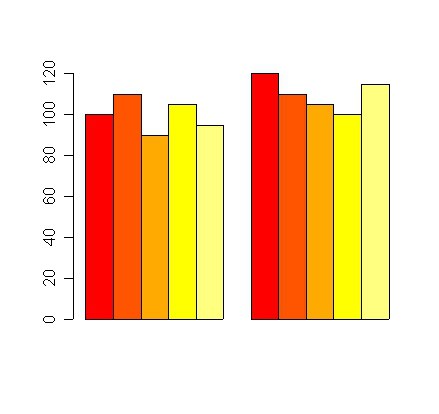
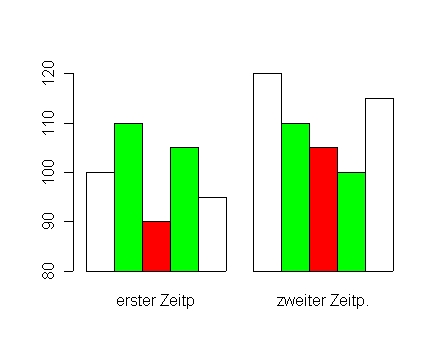
# Gruppierung besser mit Matrix als Input # beside=T bewirkt, daß die Einzelwerte einer Gruppe als einzelne Säule der Gruppe auftauchen # jede Spalte der Datenmatrix ist eine Säulengruppe dd <- cbind(c(100,110,90,105,95),c(120,110,105,100,115)) barplot(dd, beside=T)
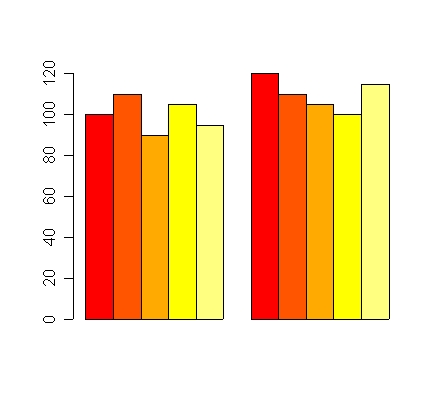
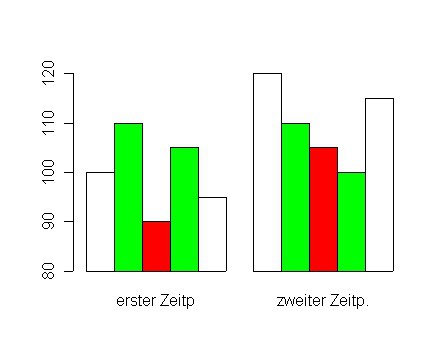
# Bars oder Bar-Gruppen beschriften
dd <- cbind(c(100,110,90,105,95),c(120,110,105,100,115))
barplot(dd,
beside=T, # Säulen nebeneinander setzen
names.arg=c('erster Zeitp','zweiter Zeitp.'), # Säulengruppen beschriften
col=c('white','green','red','green','white'), # Farben festlegen, wird für Gesamtzahl der Säulen wiederholt
ylim=c(80,120), # Y-Achsenausdehnung festlegen
xpd=FALSE # nicht über Achsen wegzeichnen
)
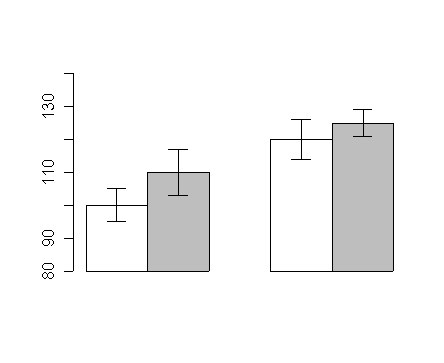
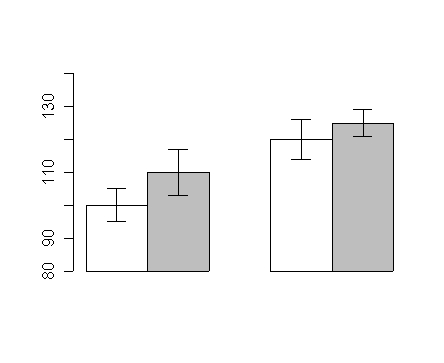
# Streuungsinformationen hinzufügen zu Barplots
# Prinzip: Pfeile hinzufügen mit 90-Grad Spitze
mw <- cbind(c(100,110),c(120,125))
se <- cbind(c(5,7),c(6,4))
xx <- barplot(mw,
beside=T, # Sääulen werden nebeneinander gezeichnet
ylim=c(80,140),
col=c('white','grey'), # die Farbenfolge wiederholt sich für die zweite Säulengruppe
xpd=F # xpd=F verhindert, daß Säulen unter die x-Achse 'sinken'
)
arrows(xx[1,1],mw[1,1],xx[1,1],mw[1,1] + se[1,1], angle=90, length=0.1)
arrows(xx[1,1],mw[1,1],xx[1,1],mw[1,1] - se[1,1], angle=90, length=0.1)
arrows(xx[2,1],mw[2,1],xx[2,1],mw[2,1] + se[2,1], angle=90, length=0.1)
arrows(xx[2,1],mw[2,1],xx[2,1],mw[2,1] - se[2,1], angle=90, length=0.1)
arrows(xx[1,2],mw[1,2],xx[1,2],mw[1,2] + se[1,2], angle=90, length=0.1)
arrows(xx[1,2],mw[1,2],xx[1,2],mw[1,2] - se[1,2], angle=90, length=0.1)
arrows(xx[2,2],mw[2,2],xx[2,2],mw[2,2] + se[2,2], angle=90, length=0.1)
arrows(xx[2,2],mw[2,2],xx[2,2],mw[2,2] - se[2,2], angle=90, length=0.1)
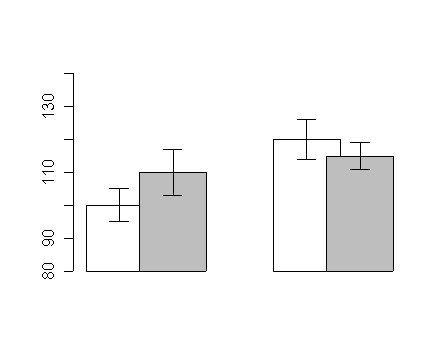
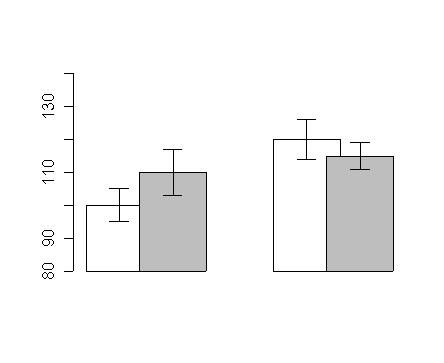
# Überlappende Balken
mw <- cbind(c(100,110),c(120,115))
se <- cbind(c(5,7),c(6,4))
xx <- barplot(mw,
beside=T,
space=c(-0.2,1), # -0.2 ist Überlappungsgrad, 1 ist Distanz zwischen Säulengruppen
ylim=c(80,140),
col=c('white','grey'),
xpd=F # xpd=F verhindert, daß Säulen unter die x-Achse 'sinken'
)
arrows(xx[1,1],mw[1,1],xx[1,1],mw[1,1] + se[1,1], angle=90, length=0.1)
arrows(xx[1,1],mw[1,1],xx[1,1],mw[1,1] - se[1,1], angle=90, length=0.1)
arrows(xx[2,1],mw[2,1],xx[2,1],mw[2,1] + se[2,1], angle=90, length=0.1)
arrows(xx[2,1],mw[2,1],xx[2,1],mw[2,1] - se[2,1], angle=90, length=0.1)
arrows(xx[1,2],mw[1,2],xx[1,2],mw[1,2] + se[1,2], angle=90, length=0.1)
arrows(xx[1,2],mw[1,2],xx[1,2],mw[1,2] - se[1,2], angle=90, length=0.1)
arrows(xx[2,2],mw[2,2],xx[2,2],mw[2,2] + se[2,2], angle=90, length=0.1)
arrows(xx[2,2],mw[2,2],xx[2,2],mw[2,2] - se[2,2], angle=90, length=0.1)
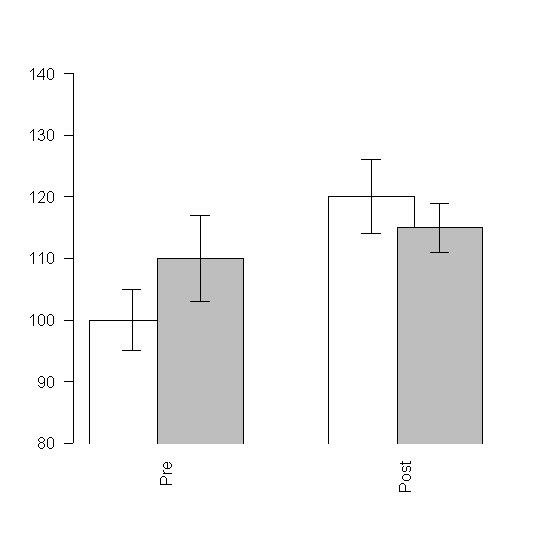
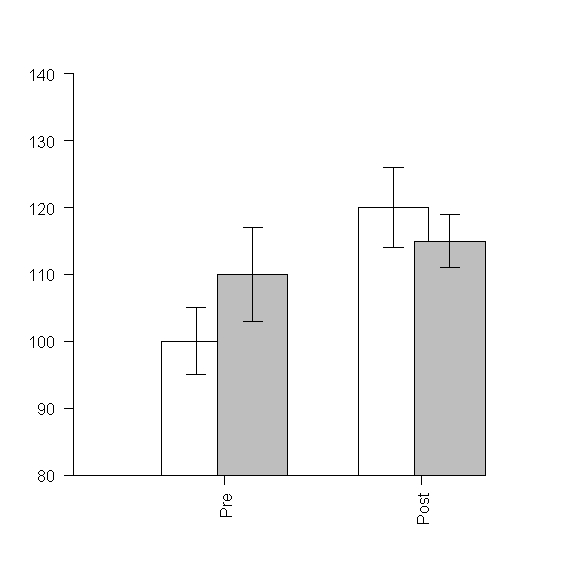
# Überlappende Balken senkrechte Beschriftung der Balken
mw <- cbind(c(100,110),c(120,115))
se <- cbind(c(5,7),c(6,4))
xx <- barplot(mw,
beside=T, # Balken werden nebeneinander geplottet
space=c(-0.2,1), # -0.2 ist Überlappungsgrad, 1 ist Distanz zwischen Säulengruppen
ylim=c(80,140), # Range der y-Achse festlegen
col=c('white','grey'), # Balkenfarben festlegen
las=2, # die Beschriftung ist bei x-Achse senkrecht (Werte zwischen 0 und 3, auch Y-Achse ist betroffen)
names.arg=c('Pre','Post'), # das steht unter den zwei Balkengruppen
xpd=F # xpd=F verhindert, daß Säulen unter die x-Achse 'sinken'
)
arrows(xx[1,1],mw[1,1],xx[1,1],mw[1,1] + se[1,1], angle=90, length=0.1)
arrows(xx[1,1],mw[1,1],xx[1,1],mw[1,1] - se[1,1], angle=90, length=0.1)
arrows(xx[2,1],mw[2,1],xx[2,1],mw[2,1] + se[2,1], angle=90, length=0.1)
arrows(xx[2,1],mw[2,1],xx[2,1],mw[2,1] - se[2,1], angle=90, length=0.1)
arrows(xx[1,2],mw[1,2],xx[1,2],mw[1,2] + se[1,2], angle=90, length=0.1)
arrows(xx[1,2],mw[1,2],xx[1,2],mw[1,2] - se[1,2], angle=90, length=0.1)
arrows(xx[2,2],mw[2,2],xx[2,2],mw[2,2] + se[2,2], angle=90, length=0.1)
arrows(xx[2,2],mw[2,2],xx[2,2],mw[2,2] - se[2,2], angle=90, length=0.1)
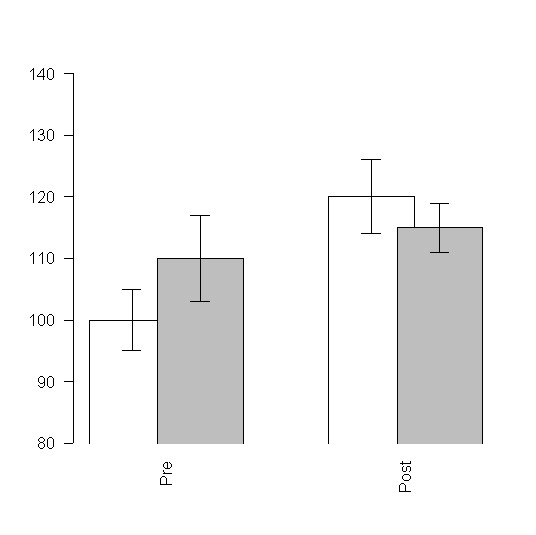
# Überlappende Balken senkrechte Beschriftung der Balken
mw <- cbind(c(100,110),c(120,115))
se <- cbind(c(5,7),c(6,4))
xx <- barplot(mw,
beside=T, # Balken werden nebeneinander geplottet
space=c(-0.2,1), # -0.2 ist Überlappungsgrad, 1 ist Distanz zwischen Säulengruppen
ylim=c(80,140), # Range der y-Achse festlegen
xlim=c(0,6),
col=c('white','grey'), # Balkenfarben festlegen
las=2, # die Beschriftung ist bei x-Achse senkrecht (Werte zwischen 0 und 3, auch Y-Achse ist betroffen)
names.arg=c('Pre','Post'), # das steht unter den zwei Balkengruppen
xpd=F, # xpd=F verhindert, daß Säulen unter die x-Achse 'sinken'
axis.lty=1 # fügt eine Linie unter den Balken ein
)
arrows(xx[1,1],mw[1,1],xx[1,1],mw[1,1] + se[1,1], angle=90, length=0.1)
arrows(xx[1,1],mw[1,1],xx[1,1],mw[1,1] - se[1,1], angle=90, length=0.1)
arrows(xx[2,1],mw[2,1],xx[2,1],mw[2,1] + se[2,1], angle=90, length=0.1)
arrows(xx[2,1],mw[2,1],xx[2,1],mw[2,1] - se[2,1], angle=90, length=0.1)
arrows(xx[1,2],mw[1,2],xx[1,2],mw[1,2] + se[1,2], angle=90, length=0.1)
arrows(xx[1,2],mw[1,2],xx[1,2],mw[1,2] - se[1,2], angle=90, length=0.1)
arrows(xx[2,2],mw[2,2],xx[2,2],mw[2,2] + se[2,2], angle=90, length=0.1)
arrows(xx[2,2],mw[2,2],xx[2,2],mw[2,2] - se[2,2], angle=90, length=0.1)
lines(c(-0.5,5.6), c(80,80)) # zieht die X-Achse wenn ylim[1] nicht 0 ist und die Balken unten offen sind
line-graphs
index
R Liniengrafiken
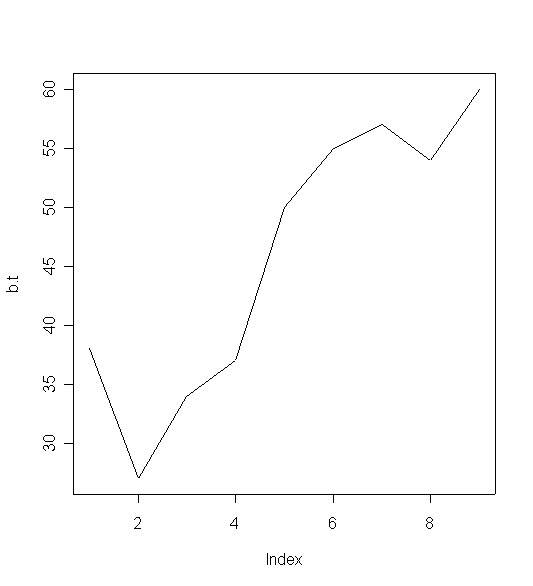
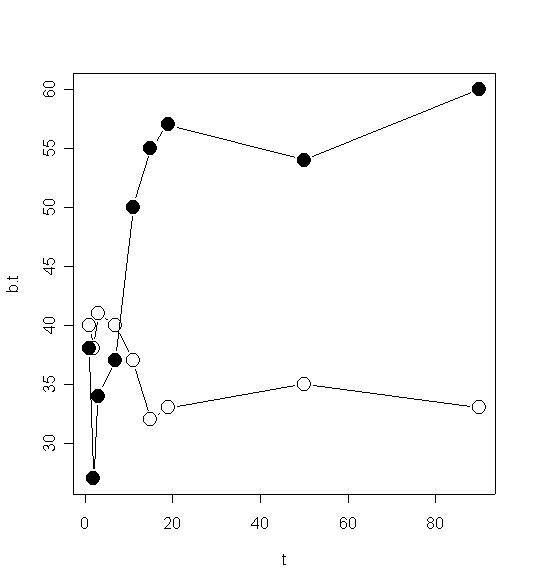
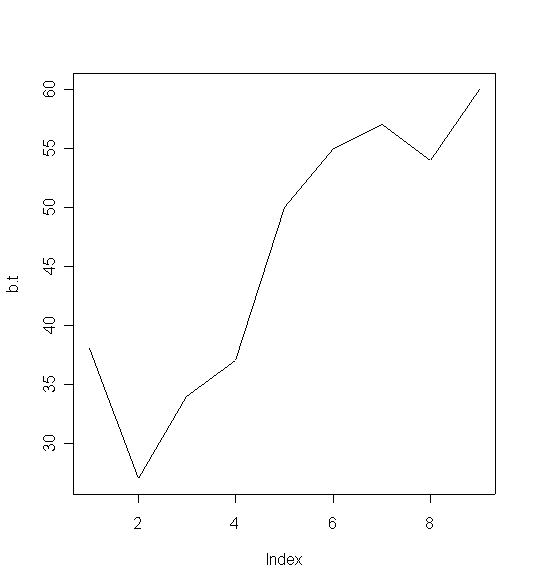
# Befinden, T-Werte (Interventions-, Kontrollgruppe, t=Messzeitpunkte) b.t <- c(38, 27, 34, 37, 50, 55, 57, 54, 60) b.w <- c(40, 38, 41, 40, 37, 32, 33, 35, 33) t <- c( 1, 2, 3, 7, 11, 15, 19, 50, 90) # Punktwolke gegen Wert-Nummer plot(b.t) # eine Linie plot(b.t, type='l')
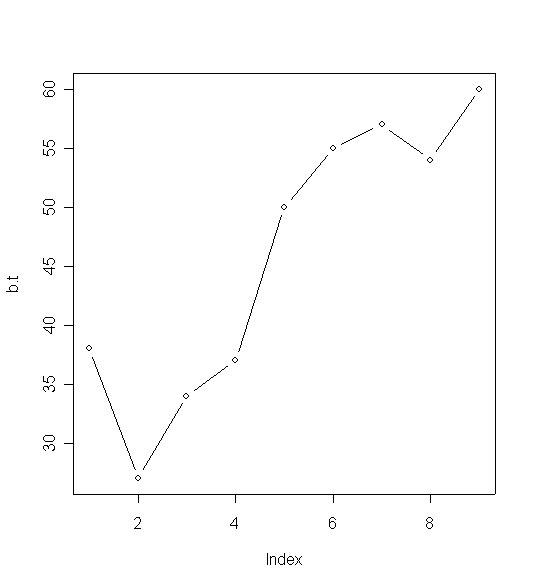
# Linie mit Symbolen plot(b.t, type='b', axes=F)
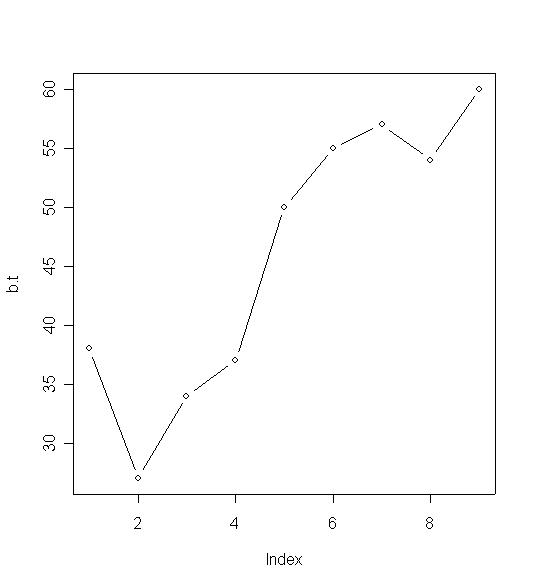
# Linie mit Symbolen, keine Default-Achsen (kommt als low-level-Befehl)
plot(b.t, type='b', axes=F)
# Default Y-Achse
axis(2)
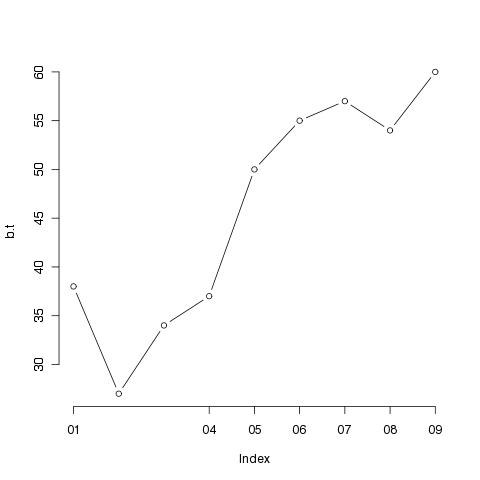
# Anpassung der X-Achse (Ticks und deren Beschriftung)
axis(1, at=c(1, 4,5,6,7,8,9), labels = c('01', '04', '05', '06', '07', '08', '09'))
# Linie mit Platz für Symbole; cex: character expansion plot(b.t, type='b', cex=0) # und die Punkte dick hinterher # lwd: line width points(b.t, lwd=4, cex=3) # dasselbe mit ausgefülltem Kreis points(b.t, lwd=3, cex=3, pch=19) # Multiple Linien, verschiedene Symbole plot(cbind(t, b.t), type='b', pch=19, cex=2) points(cbind(t, b.w), type='b', pch=21, cex=2)
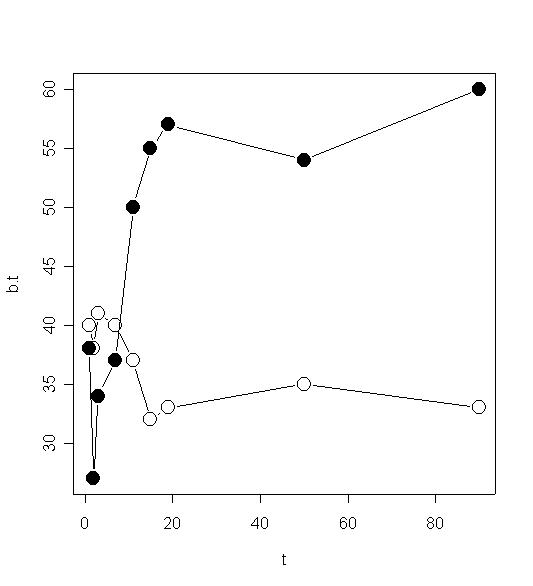
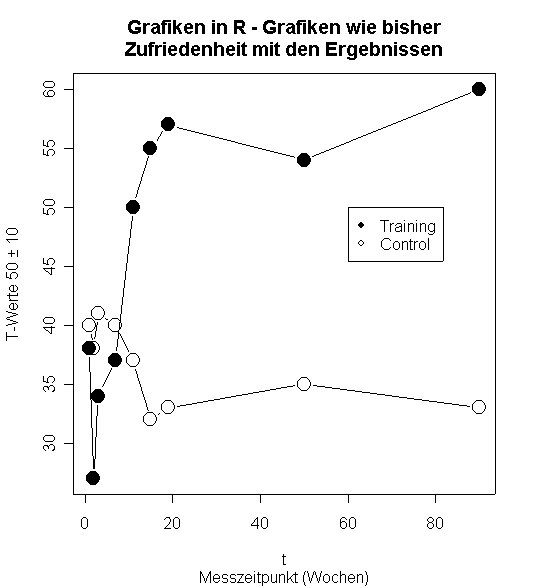
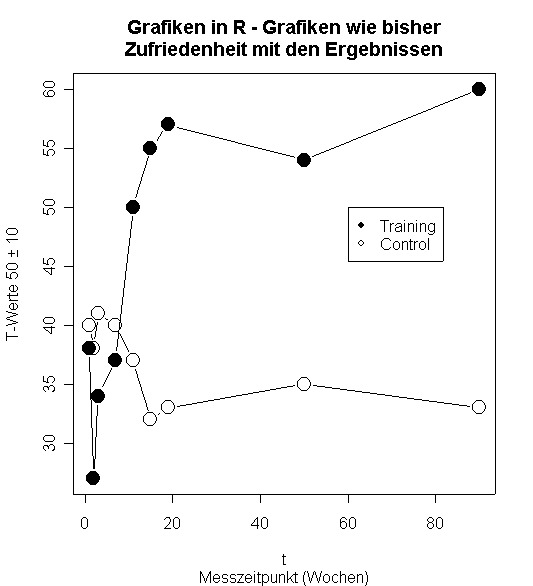
# Anhübschen
plot(cbind(t, b.t), type='b', pch=19, cex=2,
main='Grafiken in R - Grafiken wie bisher\nZufriedenheit mit den Ergebnissen',
ylab='T-Werte 50 ± 10',
sub='Messzeitpunkt (Wochen)'
)
legend(60, 50, c('Training','Control'), pch=c(19, 21))
points(cbind(t, b.w), type='b', pch=21, cex=2)
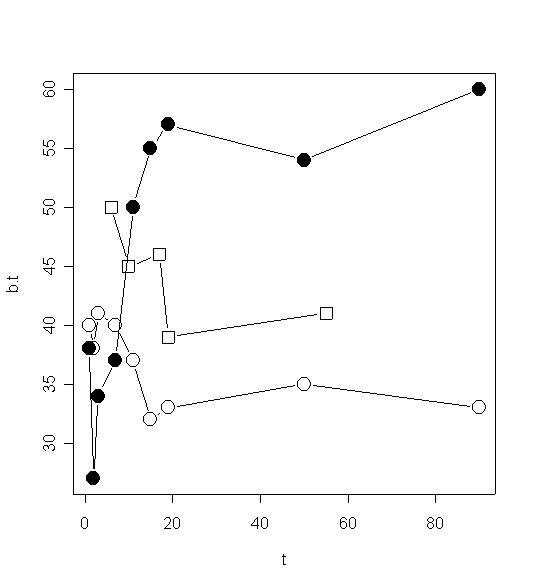
# Linien unterschiedlicher Länge b.t <- c(38, 27, 34, 37, 50, 55, 57, 54, 60) b.w <- c(40, 38, 41, 40, 37, 32, 33, 35, 33) t <- c( 1, 2, 3, 7, 11, 15, 19, 50, 90) b.2 <- c(50, 45, 46, 39, 41) t.2 <- c( 6, 10, 17, 19, 55) plot(cbind(t, b.t), type='b', pch=19, cex=2) points(cbind(t, b.w), type='b', pch=21, cex=2) points(cbind(t.2, b.2), type='b', pch=22, cex=2)
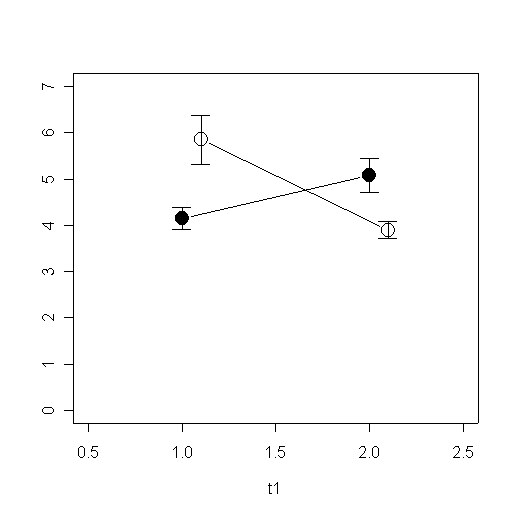
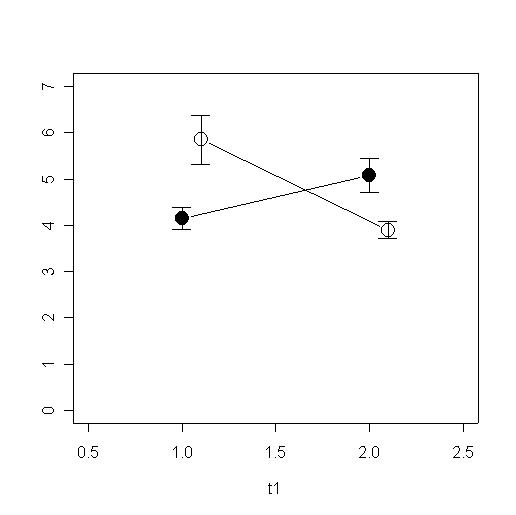
# Zwei Linien mit Streuungsbalken mw <- cbind(c(4.15,5.08),c(5.85,3.9)) se <- cbind(c(0.89 / sqrt(13), 1.32 / sqrt(13)),c(1.89 / sqrt(13),0.57 / sqrt(10))) t1 <- c(1, 2) t2 <- c(1.1, 2.1) plot(cbind(t1, mw[,1]), type='b', pch=19, cex=2, ylim=c(0,7), xlim=c(0.5,2.5)) points(cbind(t2, mw[,2]), type='b', pch=21, cex=2) arrows(t1[1],mw[1,1],t1[1],mw[1,1] + se[1,1], angle=90, length=0.1) arrows(t1[1],mw[1,1],t1[1],mw[1,1] - se[1,1], angle=90, length=0.1) arrows(t1[2],mw[2,1],t1[2],mw[2,1] + se[2,1], angle=90, length=0.1) arrows(t1[2],mw[2,1],t1[2],mw[2,1] - se[2,1], angle=90, length=0.1) arrows(t2[1],mw[1,2],t2[1],mw[1,2] + se[1,2], angle=90, length=0.1) arrows(t2[1],mw[1,2],t2[1],mw[1,2] - se[1,2], angle=90, length=0.1) arrows(t2[2],mw[2,2],t2[2],mw[2,2] + se[2,2], angle=90, length=0.1) arrows(t2[2],mw[2,2],t2[2],mw[2,2] - se[2,2], angle=90, length=0.1)
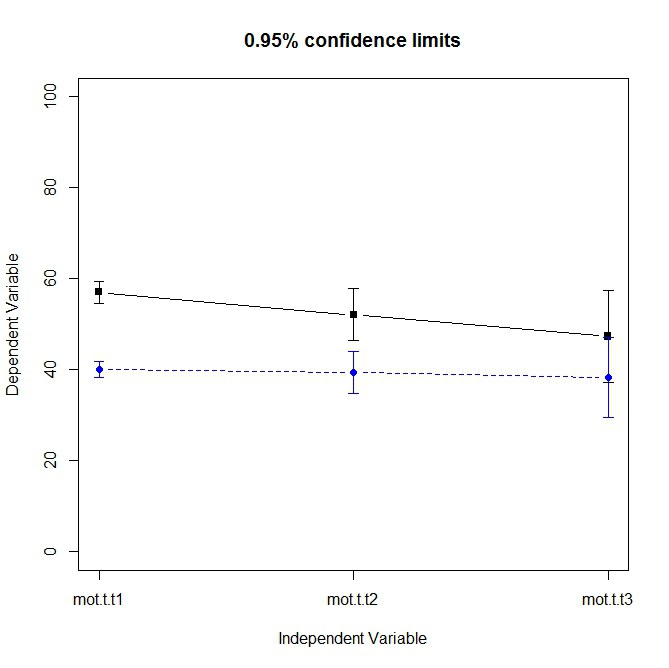
Alternativ und mit weniger Aufwand: Benutzen des Paketes psych
library(psych)
dd <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/virtual-motoric.txt")
dd[1:5,]
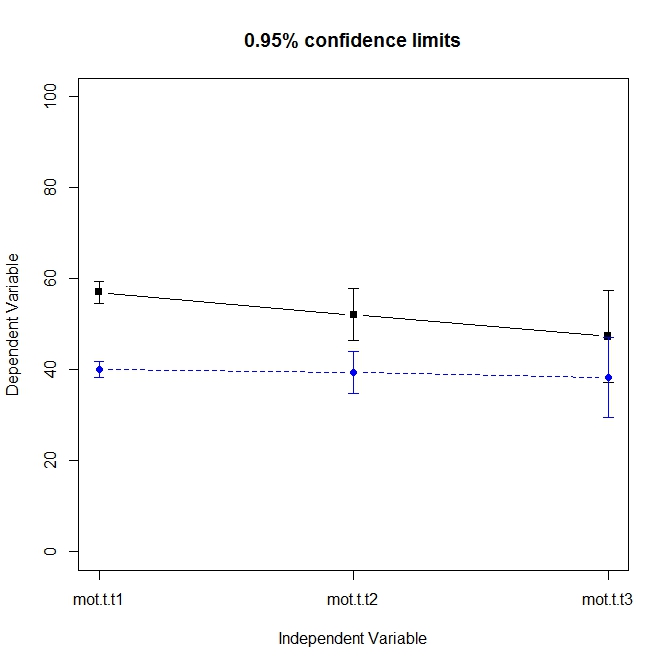
# error.bars.by library(psych)
error.bars.by(dd[,3:5], dd[,2] < 50, ylim=c(0, 100))
regression_simple
index
Einfache Regression
Ein paar Basics
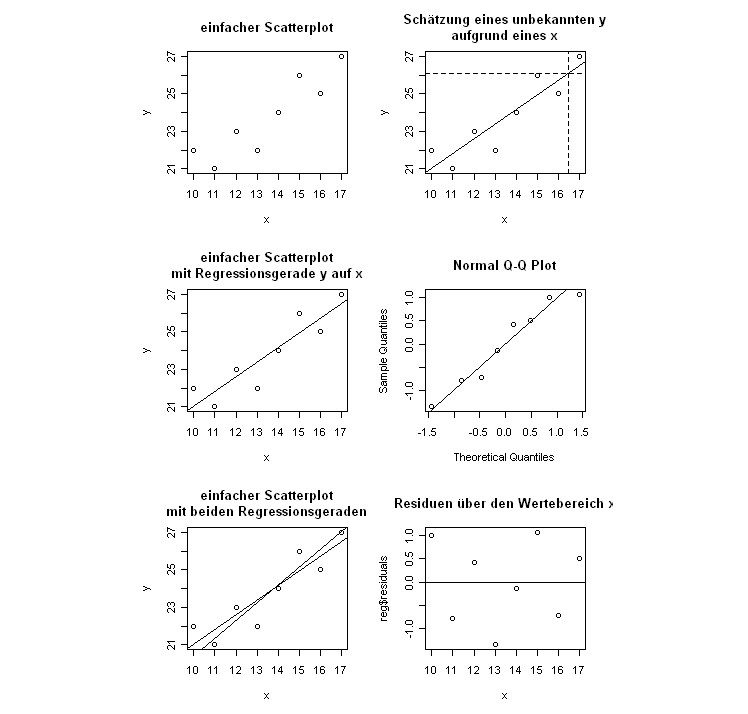
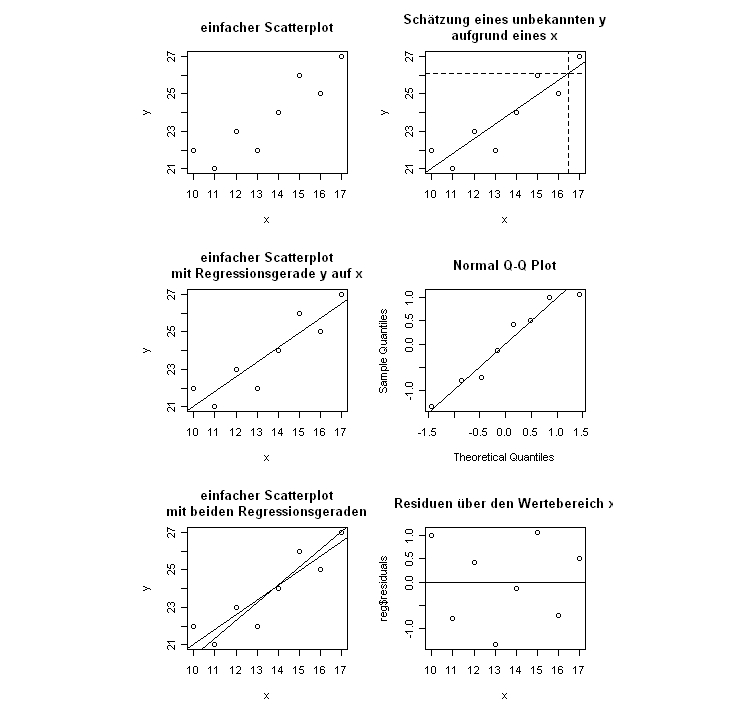
# einfache Regression # Produkt Moment Korrelation x <- c(10,11,12,13,14,15,16,17) y <- c(22,21,23,22,24,26,25,27) cor(x,y) # Visualisierung Scatterplot x <- c(10,11,12,13,14,15,16,17, 15, 17, 10) y <- c(22,21,23,22,24,26,25,27, 22, 22, 15) plot(x, y) # oder mit Nullpunkt (veränderte Achsen) plot(x, y, ylim=c(-5,30), xlim=c(-5,20)) abline(h=0) abline(v=0) #? Regression: Vorhersage der y bei gegebenen x model <- lm(y ~ x) # die Koeffizienten (Y-Achsen-Abschnitt, Steigung) model # einzeichnen abline(model) # Zweite Regressionsgerade einzeichnen # Modell Rechnen ymodel <- lm(x ~ y) # Koeffizienten ymodel # Die Koeffizienten wären nur direkt einsetzbar, wenn man die x- und y-Achsen austauschen würde. # Durch Umrechnen kann man sie in den bestehenden Plot einfügen. # Constant (y-Achsenabschnitt): Bezogen auf die bestehende Grafik gibt y.c den x-Wert an, bei dem y = 0 # Ansatz: y = y.s * x + y.c # für y = 0 # 0 = y.s * x + y.c # auflösen nach x # 0 - y.c = y.s * x # (0 - y.c)/ y.s = x # da in aktueller Grafik y und x vertauscht sind, ist dies der y-Achsenabschnitt y.c <- ymodel$coefficients[1] # Steigung: 1 / y.s # y.s sind x-Einheiten pro 1 y für den bestehenden Plot daher Umrechnung nötig. y.s <- ymodel$coefficients[2] print(c(y.c, y.s)) abline((0 - y.c) / y.s, 1/y.s, lty=4) # alles was ich über oben gerechnete Regression erfahren kann attributes(model) # alternativ nur die Namen der Attribute des 'Auswertungs-Objektes' names(model) # Zugriff über objekt['attribut-name'] model['fitted.values'] # vorhergesagte Werte model$fitted.values # alternativer Zugriff # z. B. die Residuen residuals(model) # alternativ natürlich model$residuals # oder die Parameter für die Geradengleichung coefficients(model) # par 1 ist Prädiktor-Achsenabschnitt, par 2 ist Steigung(Multiplikator für Kriterium) # Abbildungen machen # sechs vergleichende Abbildungen neben- bzw übereinander vorbereiten: nf<-layout(matrix(c(1,4,2,5,3,6),3,2,byrow=TRUE),c(1,1),c(1,1),TRUE) # Siehe /r/graph/multiple reg <- lm(y ~ x) plot(y ~ x, main='einfacher Scatterplot') plot(y ~ x, main='einfacher Scatterplot\nmit Regressionsgerade y auf x') abline(reg) plot(y ~ x, main='einfacher Scatterplot\nmit beiden Regressionsgeraden') abline(reg) ylm <- lm(x ~ y) yc <- coefficients(ylm) abline((0 - yc[1])/yc[2], 2 - yc[2]) # Schätzung für ein x = 16.5 plot(y ~ x, main='Schätzung eines unbekannten y\naufgrund eines x') abline(reg) abline(v=16.5, lty=2) # den Schätzwert von y einzeichnen abline(h=reg$coefficients[1] + reg$coefficients[2] * 16.5, lty=2) # Normalverteilung der Residuen qqnorm(reg$residuals) # wenn man möchte kann man auch die Winkelhalbierende als Gerade durchlegen abline(c(0,0), c(1,1)) # alternativ abline(0,1) # Plot der Residuen gegen die Prädiktoren plot(x, reg$residuals, main='Residuen über den Wertebereich x') abline(h=0) # Plot der Residuen gegen die vorhergesagten Werte (predicted values) #plot(reg$fitted.values, reg$residuals, main='Residuen über predicted values x')
Ein Prädiktor
Plot machen, Regressionsgerade fitten, einzeichnen, 95% Konfidenzintervall anzeigen, Modellparameter ansehen.
# Daten einlesen
dlm <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/virtual-lm.txt")
# Grafikumgebung reset
par(mfrow=c(1,1))
# zwei vergleichende Abbildungen übereinander vorbereiten:
nf<-layout(matrix(c(1,2,4,3),2,2,byrow=TRUE),c(1,1),c(1,1),TRUE)
# der Standardweg
# d2 durch d1 vorhersagen
# Modell rechnen und das Ergebnisobjekt speichern
reg <- lm(dlm$d2 ~ dlm$d1)
# ein erster Plot. Achtung: Im Plot-Befehl wird auch das Modell angegeben
plot(dlm$d2 ~dlm$d1)
# das hat den Vorteil, dass auch die Regressionsgerade einzeichnen dem Modell entnommen werden kann
abline(reg)
# Sind die Residuen normalverteilt? grafische Überprüfung.
qqnorm(reg$residuals,main="NV der Residuen?")
# Residuen ansehen
plot(dlm$d1, reg$residuals, main="Residuen gegen Prädiktor")
# und die Null-Linie
abline(h=0)
# Residuen gegen vorhergesagte Y-Werte
#plot(reg$fitted.values, reg$residuals, main="Residuen gegen Prädiktor")
# oder äquivalent mit Model-Schreibweise
#plot(reg$residuals ~ reg$fitted.values, main="Residuen gegen Prädiktor2")
# Grundplot
plot(dlm$d2 ~dlm$d1)
abline(lm(dlm$d2 ~dlm$d1))
# Berechnung der Standardfehler der Schätzung
# predict errechnet vorhergesagte Werte für Modelle
# se.fit ist ein Flag, das die Streuung des Vorhersagefehlers mit berechnet
# alles wird im Auswertungsobjekt pred gespeichert
pred<-predict(reg, se.fit=TRUE)
fitval <- pred$fit
se <- pred$se.fit
# Vorbereitung für die Visualisierung der SE-Verläufe
# den Prädiktor in einen Datenvektor 'index' speichern, der die Positionen der d1-Werte enthält, wenn sie der Größe nach geordnet werden
index <- order(dlm$d1)
# die Werte müssen der Größe nach geordnet geplottet werden, damit die Linien nicht hin- und herspringen und ein Knäuel in der Abbildung entsteht.
y <- fitval[index] # die zugehörigen vorhergesagten Werte ermitteln
stde <- se[index] # die zugehörigen Standardfehler ermitteln
yu<-y+1.96*stde # einen Datenvektor mit den Obergrenzen des Konfidenzintervalls bauen
yl<-y-1.96*stde # dasselbe mit den Untergrenzen
# und nun die beiden Verläufe einzeichnen
lines(dlm$d1[index],yu,lty=2)
lines(dlm$d1[index],yl,lty=2)
Ein Prädiktor aber mehrere Komponenten davon
In ein Modell kann zusätzlich zu der linearen Komponente auch eine quadratische oder höhere Komponente ein- und desselben Prädiktors hinzugefügt werden.
# Daten einlesen
lm.data <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/virtual-lm.dat")
# welche Spalten/Variablen gibt es und wie heissen sie?
names(lm.data)
# ein erster grafischer Überblick über die Verhältnisse
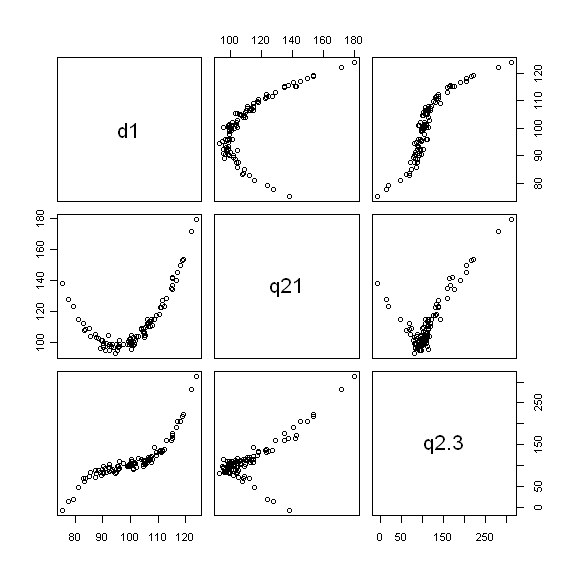
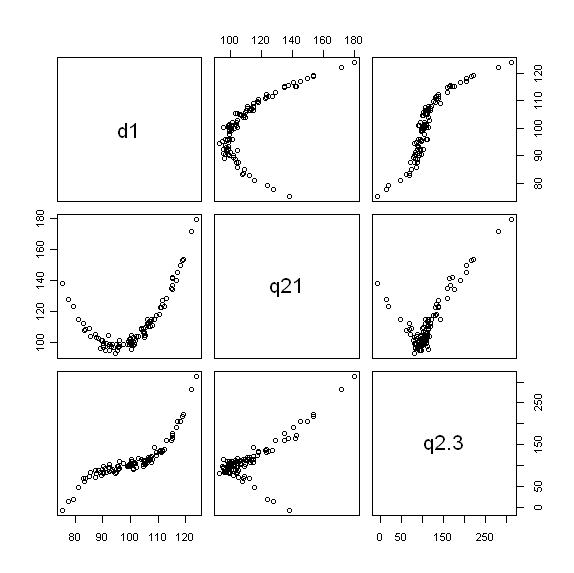
pairs(lm.data[c('d1','q21', 'q2.3')])
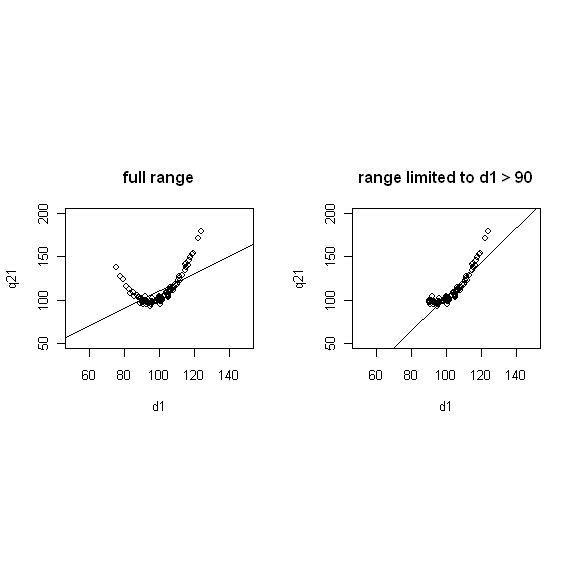
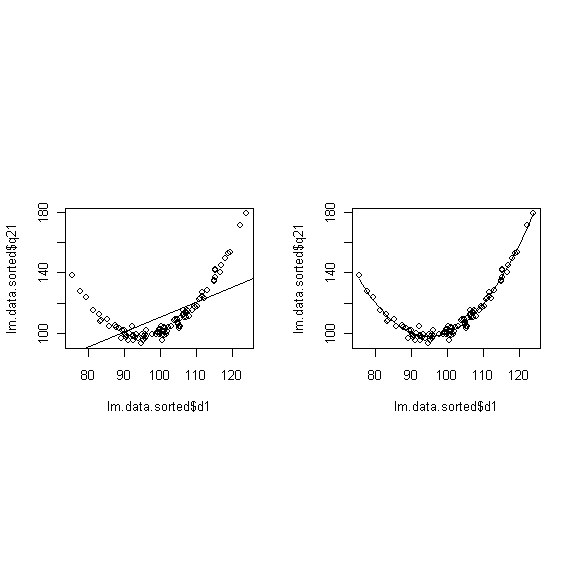
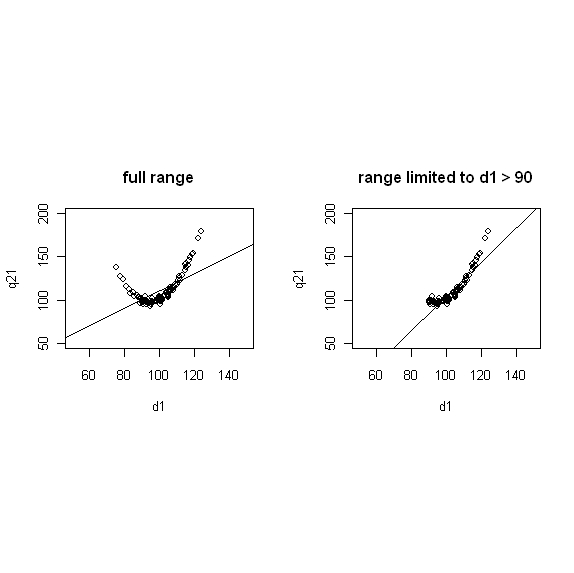
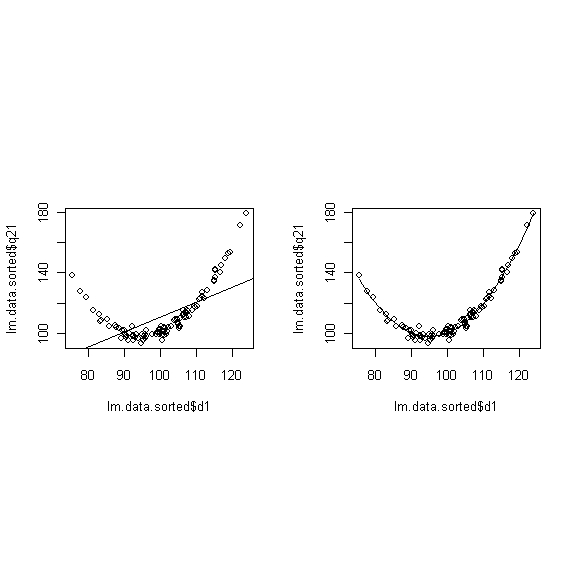
# zwei vergleichende Abbildungen übereinander vorbereiten: nf<-layout(matrix(c(1,2,0,0),2,2,byrow=TRUE),c(2,1),c(2,2),TRUE) # zwei vergleichende Abbildungen nebeneinander vorbereiten (vertikal gestreckt): par(mfrow = c(1, 2)) # zwei vergleichende Abbildungen nebeneinander vorbereiten: nf<-layout(matrix(c(1,2,0,0),2,2,byrow=TRUE),c(2,2),c(2,0),TRUE) # vier vergleichende Abbildungen neben- bzw übereinander vorbereiten: nf<-layout(matrix(c(1,2,0,0),2,2,byrow=TRUE),c(2,2),c(2,0),TRUE) # q21 über d1 vorhersagen # simple linear model r.l <- lm(lm.data$q21 ~lm.data$d1) summary(r.l) plot(lm.data$d1, lm.data$q21, ylim=c(50,200), xlim=c(50,150), xlab="d1", ylab="q21", main="full range") # und die Regressionsgerade dazu abline(lm(r.l)) # eine angepasste Linie kann täuschend gut sein # nur die d1 > 90 berücksichtigen xx <- lm.data$d1[lm.data$d1 > 90] yy <- lm.data$q21[lm.data$d1 > 90] rr <- lm(yy ~xx) summary(rr) plot(xx, yy, ylim=c(50,200), xlim=c(50,150), xlab="d1", ylab="q21", main="range limited to d1 > 90") # und die Regressionsgerade dazu abline(rr)
# eine quadratische Komponente von d1 mit dazu nehmen # zwei vergleichende Abbildungen nebeneinander vorbereiten: nf<-layout(matrix(c(1,2,0,0),2,2,byrow=TRUE),c(2,2),c(2,0),TRUE) # für eine vernünftige Darstellung muss die Tabelle nach d1 sortiert werden lm.data.sorted <- lm.data[order(lm.data$d1),] dq <- lm.data.sorted$d1^2 # Das Modell rechnen lassen r.l <- lm(lm.data.sorted$q21 ~lm.data.sorted$d1) r.lq <- lm(lm.data.sorted$q21 ~lm.data.sorted$d1 + dq) # Wie sehen die Parameter aus? summary(r.lq) # und zwei Abbildungen dazu # Abb 1, nur Linie als Modell plot(lm.data.sorted$q21 ~lm.data.sorted$d1) abline(lm(r.l)) # Abb2, quadratische Komponente hinzugenommen plot(lm.data.sorted$q21 ~lm.data.sorted$d1) lines(lm.data.sorted$d1,r.lq$fit)
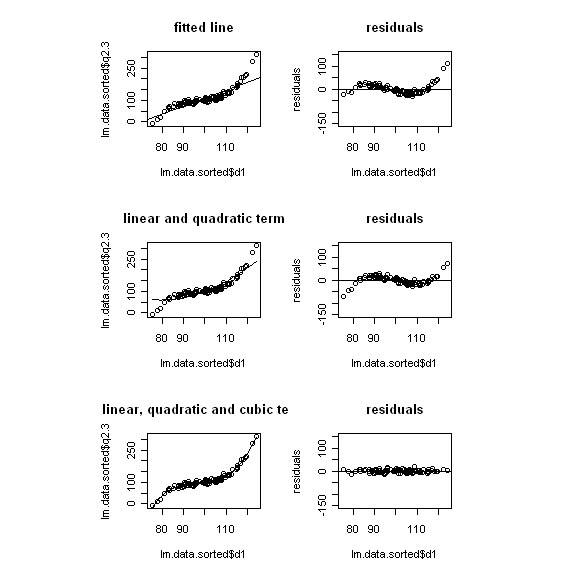
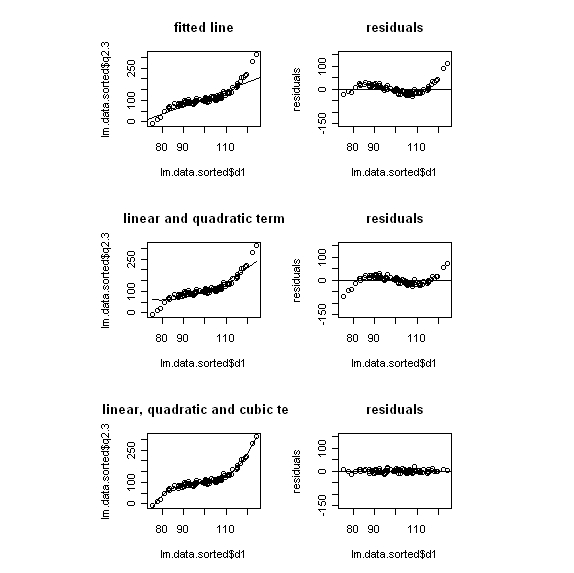
# eine lineare, eine quadratische und eine cubische Komponente von d1 mit dazu nehmen # vergleichende Abbildungen nebeneinander vorbereiten: nf<-layout(matrix(c(1,2,3,4,5,6),3,2,byrow=TRUE),c(1,1,1),c(1,1),TRUE) # für eine vernünftige Darstellung muss die Tabelle nach d1 sortiert werden lm.data.sorted <- lm.data[order(lm.data$d1),] dq <- lm.data.sorted$d1^2 dc <- lm.data.sorted$d1^3 # Das Modell rechnen lassen r.l <- lm(lm.data.sorted$q2.3 ~lm.data.sorted$d1) r.lq <- lm(lm.data.sorted$q2.3 ~lm.data.sorted$d1 + dq) r.lqc <- lm(lm.data.sorted$q2.3 ~lm.data.sorted$d1 +dq + dc) # Wie sehen die Parameter aus? summary(r.l) summary(r.lq) summary(r.lqc) # und Abbildungen dazu # Abb 1, nur Linie als Modell plot(lm.data.sorted$q2.3 ~ lm.data.sorted$d1, main="fitted line") abline(lm(r.l)) # Residuen dazu plot(r.l$residuals ~lm.data.sorted$d1, ylab="residuals", ylim=c(-150,150), main="residuals") abline(h=0) # Abb2, quadratische Komponente hinzugenommen plot(lm.data.sorted$q2.3 ~lm.data.sorted$d1, main="linear and quadratic term") lines(lm.data.sorted$d1,r.lq$fit) # Residuen dazu plot(r.lq$residuals ~lm.data.sorted$d1, ylab="residuals", ylim=c(-150,150), main="residuals") abline(h=0) # Abb3, cubische Komponente hinzugenommen plot(lm.data.sorted$q2.3 ~lm.data.sorted$d1, main="linear, quadratic and cubic term") lines(lm.data.sorted$d1,r.lqc$fit) # Residuen dazu plot(r.lqc$residuals ~lm.data.sorted$d1, ylab="residuals", ylim=c(-150,150), main="residuals") abline(h=0)
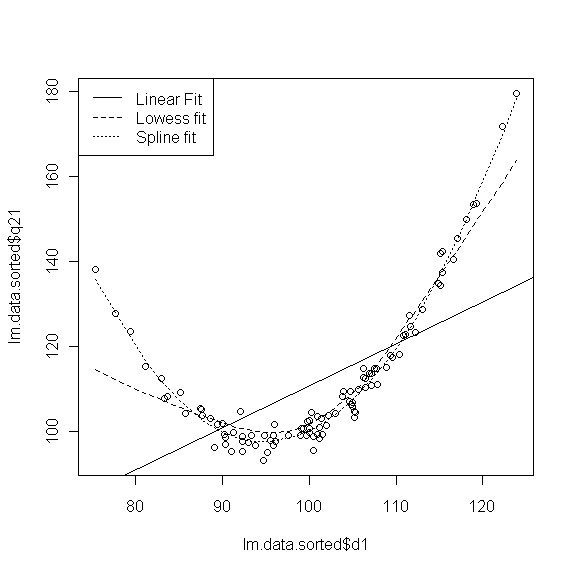
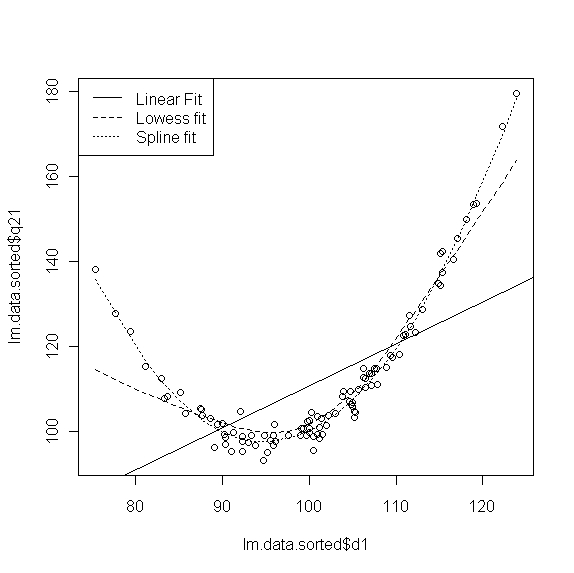
Ein Modell mit Hilfe von Smoothing-Techniken (Spline) anpassen.
Hier handelt es sich nicht um ein mathematisches Modell des Zusammenhanges von Prädiktor und Kriterium, sondern um eine grafische Techniken (Glättung). Hier kurz angerissen sind "lowess-fit" und "spline".
# Spline anpassen
# Grafikumgebung reset
par(mfrow=c(1,1))
plot(lm.data.sorted$q21 ~ lm.data.sorted$d1)
abline(lm(lm.data.sorted$q21 ~ lm.data.sorted$d1))
lines(lowess(lm.data.sorted$q21 ~ lm.data.sorted$d1),lty=2)
lines(smooth.spline(lm.data.sorted$d1, lm.data.sorted$q21),lty=3)
legend("topleft",c("Linear Fit","Lowess fit","Spline fit"),lty=1:3)
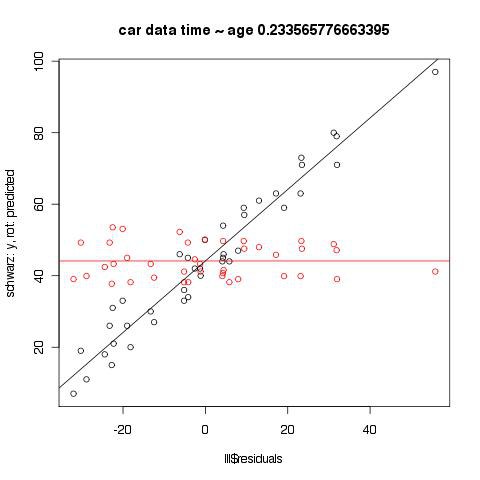
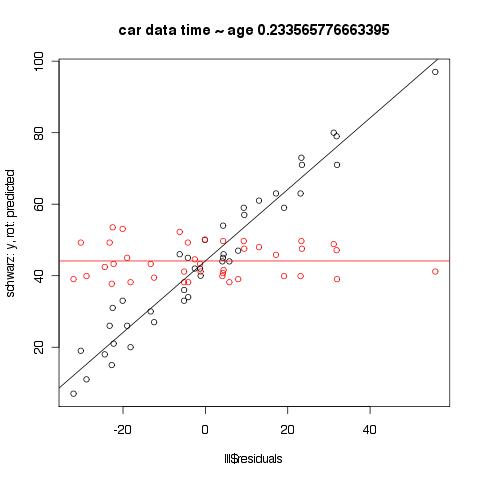
Zur Frage des Unterschiedes zwischen Residualplot geschätzte Werte vs reale Werte
Der Plot der Residuen gegen die geschätzten Werte ist geeigneter, um Hinweise auf Besonderheiten in den Residuen zu bekommen, die z. B. ein anderes Modell nahelegen könnten. Man kann im Plot unten schön den Effekt der Regression zur Mitte sehen, der bei den geschätzten Werten nicht vorhanden ist.
data <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/everitt-car-data.txt")
lll <- lm(data$time ~ data$age)
plot(lll$residuals, data$time,
main = paste("car data time ~ age", cor(data$time, data$age), sep=" ", collapse = NULL),
ylab='schwarz: y, rot: predicted')
points(cbind(lll$residuals, lll$fitted.values), type='p', pch=21, col="red")
ll.res <- lm(lll$fitted.values ~ lll$residuals)
abline(ll.res, col="red")
ll.res <- lm(data$time ~ lll$residuals)
abline(ll.res)
-
regression_multiple
index
Multiple Regression
Beispiel: Everitt (2010) car cleaning data
Daten einlesen, erster grafischer Eindruck
# Daten einlesen
car.data <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/everitt-car-data.txt")
# variablen direkt in namespace holen
attach(car.data)
# einen Eindruck der Daten bekommen (erste 10 Zeilen)
car.data[1:10,]
# eine Korrelationsmatrix
cor(car.data)
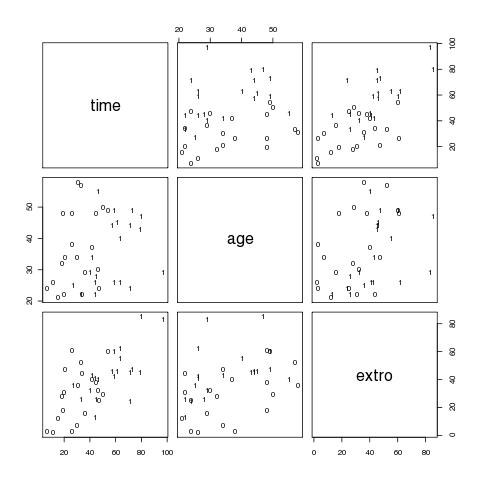
# ein erster grafischer Überblick (Fig 4.1)
# pairs erzeugt multiple scatterplots
# panel legt den Inhalt der einzelnen Fenster fest
# text trägt die Werte in der Spalte sex an die jeweils geplotteten x-y-Koordinaten aus den übergebenen Matrix-Spalten
# die 0 und 1 zeigen die Lage der verschiedenen AVen für Frauen bzw Männer
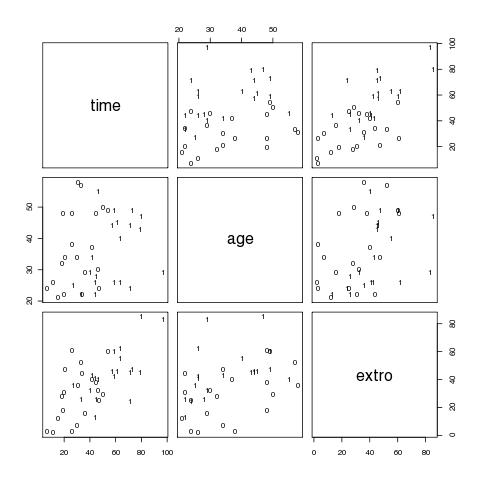
pairs(cbind(time,age,extro),panel=function(x,y) text(x,y,sex))
Linear Model spezifizieren, Anpassung rechnen lassen
# mit den drei Prädiktoren sex, age und extro
# Kurzergebnis ausgeben lassen
summary(lm(time~sex+age+extro))
# Summary zwei Prädiktoren sex, extro
summary(lm(time~sex+extro))
# ergibt output
Call:
lm(formula = time ~ sex + extro)
Residuals:
Min 1Q Median 3Q Max
-26.193 -9.474 -2.149 10.165 23.918
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 15.6797 4.4365 3.534 0.001118 **
sex 19.1801 4.4727 4.288 0.000124 ***
extro 0.5093 0.1151 4.423 8.24e-05 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 12.95 on 37 degrees of freedom
Multiple R-squared: 0.632, Adjusted R-squared: 0.6121
F-statistic: 31.77 on 2 and 37 DF, p-value: 9.284e-09
# Omnibus F-Test (F-statistic) prüft Hyp., dass alle Regressionskoeffizienten gleich 0 sind.
# bzw. ob durch die Vorhersage-Linearkombination ein signifikanter Anteil der Varianz am Kriterium erklärt wird.
# Multiple R-squared: Anteil der Varianz des Kriteriums, der durch die Kombination der Prädiktoren gebunden/erklärt wird.
# Adjusted R-squared: rechnet R-squared so um, dass die Anzahl der erklärenden Terme im Modell berücksichtigt wird.
# adjusted R-squared steigt im Gegensatz zu R-squared nur, wenn der neue Term das Modell um mehr als durch Zufall erwartet verbessert.
# adjusted R-squared kann negativ sein und ist immer <= R-squared.

p Anzahl der Regressore im linear Model (ohne constant), n ist sample-size.
# unter Coefficients finden sich die unstandardisierten Beta-Gewichte mit ihrem Standardfehler sowie ein T-Test zur Prüfung des Einzeleffekts # (t-Werte sind Estimate / Std. Error)
Vorhersage eines einzelnen Kriteriumswertes (Vp 3) mit Prädiktor sex und extero (zu Fuß)
# lm-Objekt speichern m.lm <- lm(time~sex+extro) # Summary-Objekt speichern s.lm <- summary(m.lm) # was gibt es im Summary-Objekt? names(s.lm) # nur die Koeffizienten brauchen wir koeff <- s.lm$coefficients[,1] # Vp 3 Vorhersagewert (fitted value) berechnen e.time.3 <- koeff['(Intercept)'] + koeff['sex'] * sex[3] + koeff['extro'] * extro[3] e.time.3 # Vorhersagewerte finden sich im lm-Objekt unter fitted.values m.lm$fitted.values # müßte gleich sein dem 3. Wert in den Vorhersagewerten m.lm$fitted.values[3]
Vorhergesagte Werte mit predict
predict(m.lm)
Standardisierte Beta-Gewichte sind vergleichbarer
# Standardisierte Beta-Gewichte: # Vars standardisieren car.data['st.sex'] <- car.data$sex / sd(car.data$sex) car.data['st.age'] <- car.data$age / sd(car.data$age) car.data['st.extro'] <- car.data$extro / sd(car.data$extro) car.data['st.time'] <- car.data$time / sd(car.data$time) # vergleichende Summaries # unstandardisiert summary(lm(car.data$time~car.data$sex+car.data$extro)) # Prädiktoren standardisiert summary(lm(car.data$time~car.data$st.sex+car.data$st.extro)) # Kriterium und Prädiktoren standardisiert summary(lm(car.data$st.time~car.data$st.sex+car.data$st.extro)) # Alternative: scale() benutzen und die Standardisierng 'on the fly' machen anstelle eine neue Variable zu bilden summary(lm(scale(car.data$time) ~ scale(car.data$sex) + scale(car.data$extro))) # Alternative: standardisierte Regressionskoeffizienten aus den unstandardisieren errechnen # standardisierte Regressionskoeffizienten lassen sich auch einfach ausrechnen # raw-coeffizient(predictor) * sd(predictor) / sd(criterion) # das unstandardisierte Modell speichern ureg <- lm(car.data$time~car.data$sex+car.data$extro) # die standardisierten Regressionskoeffizienten berechnen und ausgeben ureg$coefficients['car.data$sex'] * sd(car.data$sex) / sd(car.data$time) ureg$coefficients['car.data$extro'] * sd(car.data$extro) / sd(car.data$time)
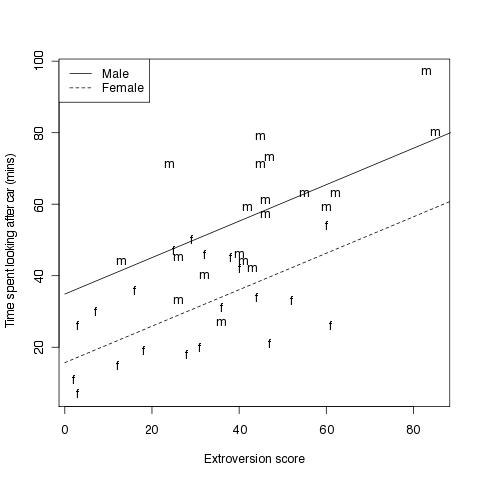
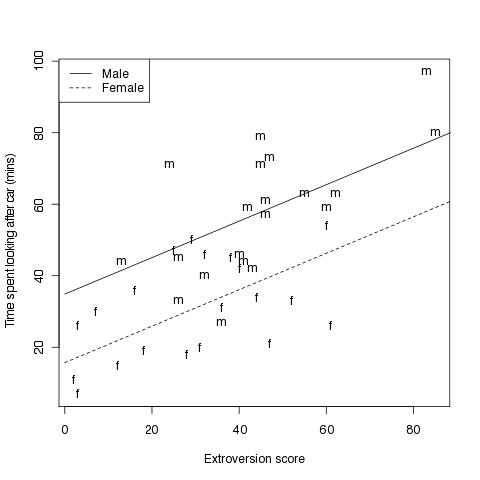
Einen Scatterplot erstellen mit den beiden Regressionsgeraden für das jeweilige Geschlecht
#Fig 4.2
# vergleichend die beiden Regressionsgeraden für Männer und Frauen
# Grafikumgebung zurücksetzen
par(mfrow=c(1,1))
# einen Vektor mit 'm' für Maänner und 'f' für Frauen erstellen
Sex<-rep("m",length(sex))
Sex[sex==0]<-"f"
# einen Vektor mit 50 Elementen erstellen die in regelmäßigen Abständen zwischen 0 und 90 liegen
x<-seq(0,90,length=50)
# die Geradengleichungen mit derselben Steigung und verschiedenem Y-Achsenabschnitt
ym<-15.68+19.18+0.51*x
yf<-15.68+0.51*x
# und nun der plot
plot(car.data$time ~ car.data$extro, # plotte Grundmodell
xlab="Extroversion score", # X-Achsen-Beschriftung
ylab="Time spent looking after car (mins)",type="n") # Y-Achsen-Beschriftung
text(car.data$extro,car.data$time,labels=Sex) # an die Koordinaten den Text (Buchstaben) ausgeben
lines(x,ym,lty=1) # Regressionsgerade für Männer einzeichnen, Linienart festlegen
lines(x,yf,lty=2) # dito für Frauen
legend("topleft",c("Male","Female"), # eine Legende in die linke, obere Ecke
lty=1:2) # zugeordneter Linientyp in der Legende, der dem der Regressionsgeraden entspricht
Wechselwirkungsterm mit in MR einbeziehen
# Daten vorbereiten
time <- car.data$time
sex <- car.data$sex
extro <- car.data$extro
# das Modell anpassen und Summary ausgeben
rr <- lm(time~sex+extro+sex:extro)
summary(rr)
# ergibt output
Call:
lm(formula = time ~ sex + extro + sex:extro)
Residuals:
Min 1Q Median 3Q Max
-24.807 -9.445 -1.677 10.477 27.183
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 20.0182 5.4560 3.669 0.000782 ***
sex 7.8178 9.5705 0.817 0.419379
extro 0.3607 0.1590 2.268 0.029430 *
sex:extro 0.3052 0.2279 1.339 0.188970
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 12.81 on 36 degrees of freedom
Multiple R-squared: 0.6495, Adjusted R-squared: 0.6203
F-statistic: 22.23 on 3 and 36 DF, p-value: 2.548e-08
# Alternativ kann dasselbe Modell angepasst werden,
# indem ein neuer Prädiktor aus dem Produkt der alten berechnet und in das Modell mit eingefügt wird
mult.sex.extro <- sex * extro
rr2 <- lm(time ~ sex + extro + mult.sex.extro)
summary(rr2)
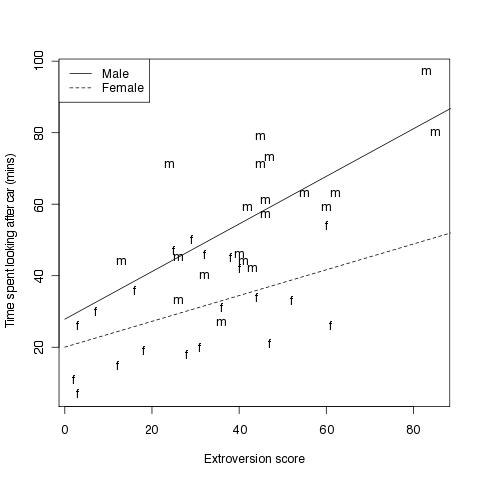
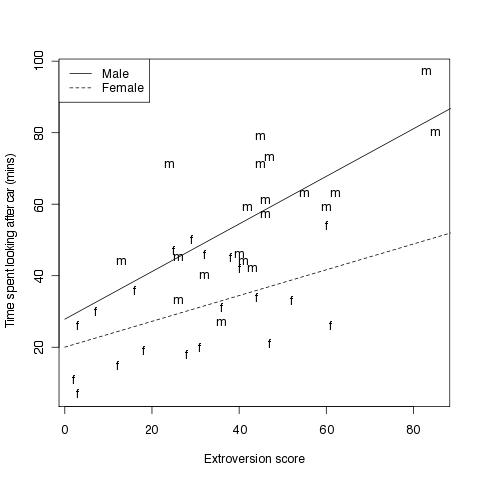
#Fig 4.3
# Abbildung die zusätzlich noch die Wechselwirkung mit einbezieht
# Grafik vorbereiten
Sex<-rep("m",length(sex))
Sex[sex==0]<-"f"
x<-seq(0,90,length=50)
# für Männer ist x = 1
#ym<-20+7.82+(0.36+0.31)*x
# eine Gerade über den Wertebereich von x mit 1
ym <- rr$coefficients['(Intercept)'] + rr$coefficients['sex'] * 1 + (rr$coefficients['extro'] * 1 + rr$coefficients['sex:extro'] * 1) * x
# für Frauen ist x = 0
# yf<-20+0.36*x
yf <- rr$coefficients['(Intercept)'] + rr$coefficients['sex'] * 0 + (rr$coefficients['extro'] * 1 + rr$coefficients['sex:extro'] * 0) * x
plot(time~extro,xlab="Extroversion score",
ylab="Time spent looking after car (mins)",type="n")
text(extro,time,labels=Sex)
lines(x,ym,lty=1)
lines(x,yf,lty=2)
legend("topleft",c("Male","Female"),lty=1:2)
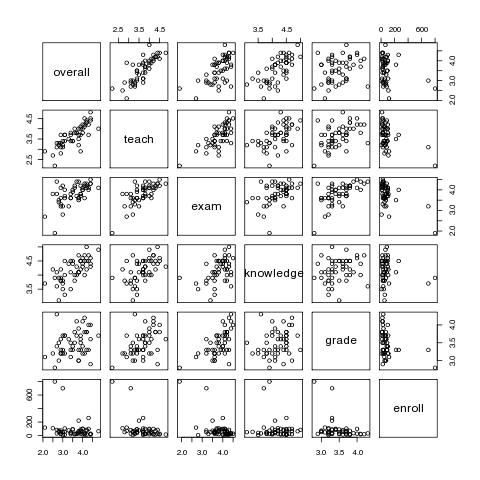
Prüfung auf Multikollinearität und Prädiktorenselektion mit Course-Data
# Daten einlesen
course.data <- read.delim("http://www.psych.uni-goettingen.de/de/mat/mv/course-data.txt")
# und die Spalten in den Namespace holen
attach(course.data)
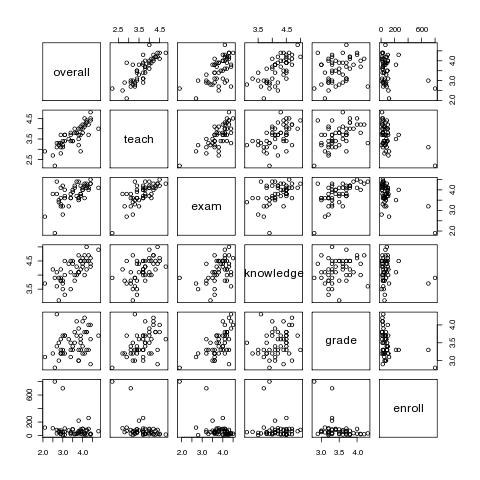
# ein Einblick in die Struktur des Data.Frames
course.data[1:10,]
# ein erster grafischer Überblick
pairs(course.data)
# oder
plot(course.data)
Kontrolle auf Probleme mit Multikollinearität mit dem VIF (Variance Inflation Factor). VIF wird gebildet aus der multiplen Korrelation eines Prädiktors mit dem Rest der Prädiktoren. Daumenregel: Wenn der VIF 10 überschreitet gibt es Probleme mit Kollinearität im Pool der Prädiktoren.
Der VIF kann leicht errechnet werden, indem man die Restprädiktoren in ein 'lm' steckt als Prädiktoren und den relevanten Prädiktor als Kriterium. Das multiple R^2 finet man im Summary.
Die Formel für VIF ist 1 / (1 - R^2)
vif.teach <- 1 /(1 - summary(lm(teach ~ exam + knowledge + grade + enroll))$r.squared) vif.teach vif.exam <- 1 /(1 - summary(lm(exam ~ teach + knowledge + grade + enroll))$r.squared) vif.exam vif.knowledge <- 1 /(1 - summary(lm(knowledge ~ teach + exam + grade + enroll))$r.squared) vif.knowledge vif.grade <- 1 /(1 - summary(lm(grade ~ teach + exam + knowledge + enroll))$r.squared) vif.grade vif.enroll <- 1 /(1 - summary(lm(enroll ~ teach + exam + knowledge + grade ))$r.squared) vif.enroll
Jede Änderung an den Prädiktoren bzw. den Vpn verändert das Gesamtgefüge und erfordert eine Neuanpassung des Modells. Das gilt bei der Herausnahme oder beim Hinzufügen eines einzigen Prädiktors oder natürlich einer Gruppe. Entscheidungen über das weitere Vorgehen müssen bei jedem Einzelschritt durch Neuanpassung überprüft werden.
forward: Anfangen mit dem Prädiktor mit der höchsten Einzelkorrelation, dann je den vielversprechendsten Prädiktor neu mit aufnehmen
backward: Anfangen mit allen Prädiktoren und dann den je 'schwächsten' aus dem Modell nehmen.
stepwise: Kombination: Schrittweise hinzunehmen und dann prüfen, ob aus den bereits integrierten Variablen sich eine als nicht mehr bedeutsam erweist.
AIC: Akaike's information criterion.
AIC 'bestraft' die Hinzunahme von Variablen, rechnet aber deren zusätzlichen Erklärungswert gegen. Frage, die das AIC beantwortet: Wie ist der Verlust an Erklärung im Vergleich mit dem Gewinn durch ein sparsameres Modell?
Kriterium: AIC soll kleiner werden (auch im Minus-Bereich)
# Backward regression # volles Modell generieren reg<-lm(overall~teach+exam+knosuwledge+grade+enroll) # backward step(reg, direction="backward") # Kriterium: AIC soll kleiner werden (auch im Minus-Bereich) # 'step' kann auch 'forward' und 'both'
Beurteilung einzelner Fälle, standardized residuals, deletion-residuals, leverage und Cook's-distance
Leverage
lever <- hatvalues(m.lm)
Cook's distance: Einfluss eines Falles auf die Parameterschätzung des Gesamtmodells. Ein Wert größer 1 heisst, dass diese Beobachtung einen unverhältnismäßig großen Einfluss hat (Empfehlung Everitt, 2010).
Die Funktion cooks.distance(), der das lineare Modell übergeben wird, liefert die cook's distance für die Beobachtungen zurück.
Ein plot() des Modells liefert mehrere Grafiken u. a. verschiedene Residual-Plots sowie einen Plot der die Cook's Distance visualisiert